Cuando el síndrome respiratorio agudo severo (SARS) surgió en el año 2003, mató al menos a 775 personas antes de ser contenido, y nueve años más tarde, el síndrome respiratorio de Medio Oriente (MERS) empezó a circular en la población humana y ha pasado a tener una tasa de letalidad de 36%.
Estas enfermedades son especies de coronavirus, que evolucionan rápidamente en patógenos que pueden “desbordarse” de las poblaciones de animales a los seres humanos. “Tenemos que estar preparados para estos virus”, dice Andrew Ward, profesor asociado en el Instituto de Investigación Scripps (TSRI).
Ahora, Ward y sus colegas en TSRI, Dartmouth y los Institutos Nacionales de Salud (NIH), todos ellos en Estados Unidos, han resuelto la estructura de una proteína clave en HKU1, un coronavirus identificado en Hong Kong en 2005 y muy relacionado con los del SARS y el MERS. Creen que sus hallazgos podrían llevar a futuros tratamientos para esta familia de virus.
“Esto es realmente el puntapié inicial. Una vez que tenga estructuras, en realidad se puede comenzar a ir después de diferentes coronavirus, como los del MERS y el SARS”, apuntó Ward, quien codirigió el estudio con Jason S. McLellan, profesor asistente de Bioquímica en la Escuela Geisel de Medicina de la Universidad de Dartmouth, Hanover, New Hampshire.
En la base de cada proteína de la espícula de los coronavirus está la maquinaria de fusión del virus, que éste utiliza para entrar en las células huésped. La estructura de esta maquinaria raramente cambia entre las especies de coronavirus, lo que significa que los anticuerpos que se dirigen a la estructura podrían funcionar contra muchos virus de esta familia.
El reto para los investigadores ha sido encontrar anticuerpos que realmente puedan llegar a la maquinaria de fusión celular. La protección de esta maquinaria viral crítica es una capa de glicoproteínas que la protege de los ataques de anticuerpos del huésped. Para ayudar a identificar formas en torno a la glicoproteína de “escudo”, los científicos necesitan un mapa de la estructura de una proteína de espícula.
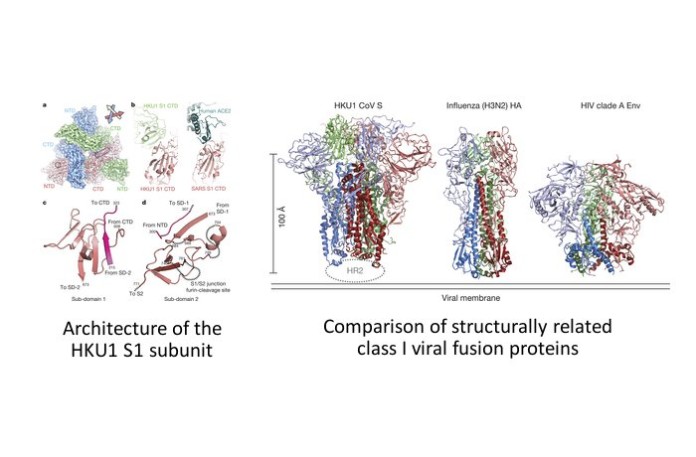
Proteínas unidas en forma de campana invertida
En el nuevo estudio, Ward y sus colegas utilizaron una técnica de imagen llamada crioelectro microscopía –en la que se congelan las muestras y luego se forma la imagen con un haz de electrones– para resolver la estructura tridimensional de la proteína espícula. Los resultados muestran que las proteínas HKU1 tienen dos lóbulos entrelazados que se entrecruzan mediante la maquinaria de fusión y construyen una especie de forma de campana invertida, una conformación no aparente en estudios previos de sólo partes de la estructura.
“Es realmente una de las primeras imágenes que tenemos de una proteína espícula de coronavirus humano”, afirma el estudiante graduado de TSRI Christopher Cottrell, que fue primer autor del trabajo junto con el investigador asociado de TSRI Robert Kirchdoerfer.
La nueva imagen estructural es también la primera en hacer alusión a la forma en que el virus reconoce los receptores de la célula huésped. Parece que la presencia de receptores celulares humanos puede hacer que la proteína de la espícula cambie su conformación y revelar su mecanismo de fusión.
Aunque el mecanismo exacto detrás del cambio todavía se desconoce, Kirchdoerfer ve este fenómeno como una vulnerabilidad que las futuras terapias podrían explotar. “Es posible inducir el cambio conformacional con un anticuerpo”, augura.
Los investigadores esperan estar preparados para un futuro evento de brote de coronavirus. “Es una buena demostración de cómo podemos aprovechar lo que hemos hecho en diferentes áreas como el VIH y el virus del Ébola para resolver rápidamente las estructuras de patógenos emergentes”, afirma Ward. El siguiente paso en esta investigación será encontrar anticuerpos humanos o animales que puedan dirigirse a los sitios de vulnerabilidad en la estructura de una proteína espícula.
Puede consultar el artículo completo, en inglés, haciendo clic aquí (requiere suscripción).
Fuente: REC